Une démonstration scientifique utilisant l’apprentissage automatique révèle des séquences d’ADN « extrêmes » avec des activités personnalisées.
L’intelligence artificielle (IA) a explosé dans nos fils d’actualité, le ChatGPT et d’autres technologies d’IA connexes devenant l’objet d’un examen public approfondi. Outre les populaires chatbots, les biologistes trouvent des moyens d’exploiter l’IA pour étudier les fonctions essentielles de nos gènes.
Des chercheurs de l’université de Californie à San Diego, qui étudient les séquences d’ADN qui activent les gènes, ont utilisé l’intelligence artificielle pour identifier une pièce énigmatique liée à l’activation des gènes, un processus fondamental impliqué dans la croissance, le développement et les maladies. Grâce à l’apprentissage automatique, un type d’intelligence artificielle, le professeur James T. Kadonaga, de l’École des sciences biologiques, et ses collègues ont découvert la région promotrice centrale en aval (DPR), un code d’activation de l’ADN « passerelle » qui intervient dans le fonctionnement de près d’un tiers de nos gènes.
Sur la base de cette découverte, Kadonaga et les chercheurs Long Vo ngoc et Torrey E. Rhyne ont utilisé l’apprentissage automatique pour identifier des séquences d’ADN synthétiques « extrêmes » ayant des fonctions spécifiquement conçues pour l’activation des gènes. Dans une publication parue dans la revue Genes & Development, les chercheurs ont testé des millions de séquences d’ADN différentes à l’aide de l’apprentissage automatique, en comparant l’élément d’activation du gène DPR chez l’homme et chez la mouche des fruits (drosophile). En utilisant l’IA, ils ont pu trouver des séquences DPR rares et personnalisées qui sont actives chez l’homme mais pas chez la drosophile, et vice versa. Plus généralement, cette approche pourrait maintenant être utilisée pour identifier des séquences d’ADN synthétiques ayant des activités qui pourraient être utiles en biotechnologie et en médecine.
« À l’avenir, cette stratégie pourrait être utilisée pour identifier des séquences d’ADN synthétiques extrêmes ayant des applications pratiques et utiles. Au lieu de comparer des humains (condition X) avec des drosophiles (condition Y), nous pourrions tester la capacité du médicament A (condition X), mais pas celle du médicament B (condition Y), à activer un gène », a déclaré M. Kadonaga, professeur émérite au département de biologie moléculaire. « Cette méthode pourrait également être utilisée pour trouver des séquences d’ADN personnalisées qui activent un gène dans le tissu 1 (condition X), mais pas dans le tissu 2 (condition Y). Les applications pratiques de cette approche basée sur l’IA sont nombreuses. Les séquences d’ADN synthétiques extrêmes peuvent être très rares, peut-être une sur un million – si elles existent, elles peuvent être trouvées grâce à l’IA »
L’apprentissage automatique est une branche de l’IA dans laquelle les systèmes informatiques s’améliorent et apprennent en permanence sur la base de données et d’expériences. Dans cette nouvelle recherche, Kadonaga, Vo ngoc (ancien chercheur postdoctoral à l’UC San Diego, aujourd’hui à Velia Therapeutics) et Rhyne (chercheur associé) ont utilisé une méthode connue sous le nom de régression vectorielle de support pour « former » des modèles d’apprentissage automatique avec 200 000 séquences d’ADN établies sur la base de données provenant d’expériences réelles en laboratoire. Il s’agissait des cibles présentées comme exemples pour le système d’apprentissage automatique. Ils ont ensuite « introduit » 50 millions de séquences d’ADN de test dans les systèmes d’apprentissage automatique pour les humains et les mouches des fruits et leur ont demandé de comparer les séquences et d’identifier les séquences uniques dans les deux énormes ensembles de données.
Bien que les systèmes d’apprentissage automatique aient montré que les séquences humaines et celles des mouches des fruits se chevauchaient largement, les chercheurs se sont concentrés sur la question centrale de savoir si les modèles d’intelligence artificielle pouvaient identifier les rares cas où l’activation des gènes est très active chez l’homme mais pas chez la mouche des fruits. La réponse a été un « oui » retentissant. Les modèles d’apprentissage automatique ont été capables d’identifier des séquences d’ADN spécifiques à l’homme (et spécifiques à la mouche à fruits). Il est important de noter que les fonctions des séquences extrêmes prédites par l’IA ont été vérifiées dans le laboratoire de M. Kadonaga à l’aide de méthodes d’essai conventionnelles.
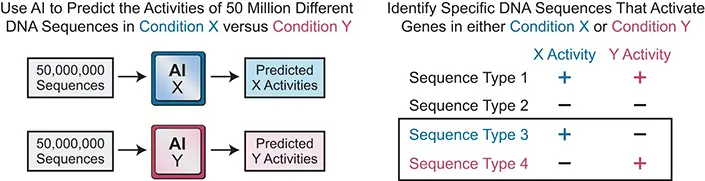
avant de nous lancer dans ce travail, nous ne savions pas si les modèles d’IA étaient suffisamment « intelligents » pour prédire les activités de 50 millions de séquences, en particulier les séquences atypiques « extrêmes » aux activités inhabituelles. Il est donc très impressionnant et tout à fait remarquable que les modèles d’IA aient pu prédire les activités des rares séquences extrêmes, une sur un million », a déclaré M. Kadonaga, ajoutant qu’il serait pratiquement impossible de réaliser les 100 millions d’expériences de laboratoire comparables que la technologie d’apprentissage automatique a analysées, car il faudrait près de trois semaines pour réaliser chaque expérience de laboratoire.
Les séquences rares identifiées par le système d’apprentissage automatique constituent une démonstration réussie et ouvrent la voie à d’autres utilisations de l’apprentissage automatique et d’autres technologies d’intelligence artificielle en biologie.
« Dans la vie de tous les jours, les gens découvrent de nouvelles applications pour les outils d’IA, tels que ChatGPT. Ici, nous démontrons l’utilisation de l’IA pour concevoir des éléments d’ADN personnalisés afin d’activer des gènes. Cette méthode devrait avoir des applications pratiques en biotechnologie et en recherche biomédicale », a déclaré M. Kadonaga. « D’une manière plus générale, les biologistes commencent probablement à peine à explorer la puissance de la technologie de l’IA
La recherche a été financée par les National Institutes of Health (R35 GM118060).
Source : UCSD


